2 Illustrationer
2.1 Histogram
Eksempel: Vitamin D (kun irske kvinder, uge 1)
Se i R-intro hvordan du lægger tæthedskurver på histogram
Bemærk at jeg her lægger normalfordelingskurven på ved brug af curve(), hvor man slipper for at beregne værdierne som ellers vist i R-intro. Med add=TRUE tvinger vi R til at lægge kurven oven i plottet (ellers får vi en helt ny figur), og skal som første argument blot angive at vi ønsker en normalfordeling (dnorm Density NORMal) beregnet for alle værdier på x-aksen (her skal vi skrive x i første argument) med middelværdi og SD svarende til data for de irske kvinder.
Brug af legends er beskrevet i afsnit om basal grafik
hist( vitEI$vitd, col='lightgray', xlab='Vitamin D', main="Irske kvinder", prob=T)
lines( density( vitEI$vitd ), lwd=2)
curve( dnorm( x, mean=mean(vitEI$vitd), sd=sd( vitEI$vitd) ),
col="red", lwd=2, add=TRUE)
legend('topright', c('datatilpasset','normalfordeling'), lty=1, col=1:2, lwd=2)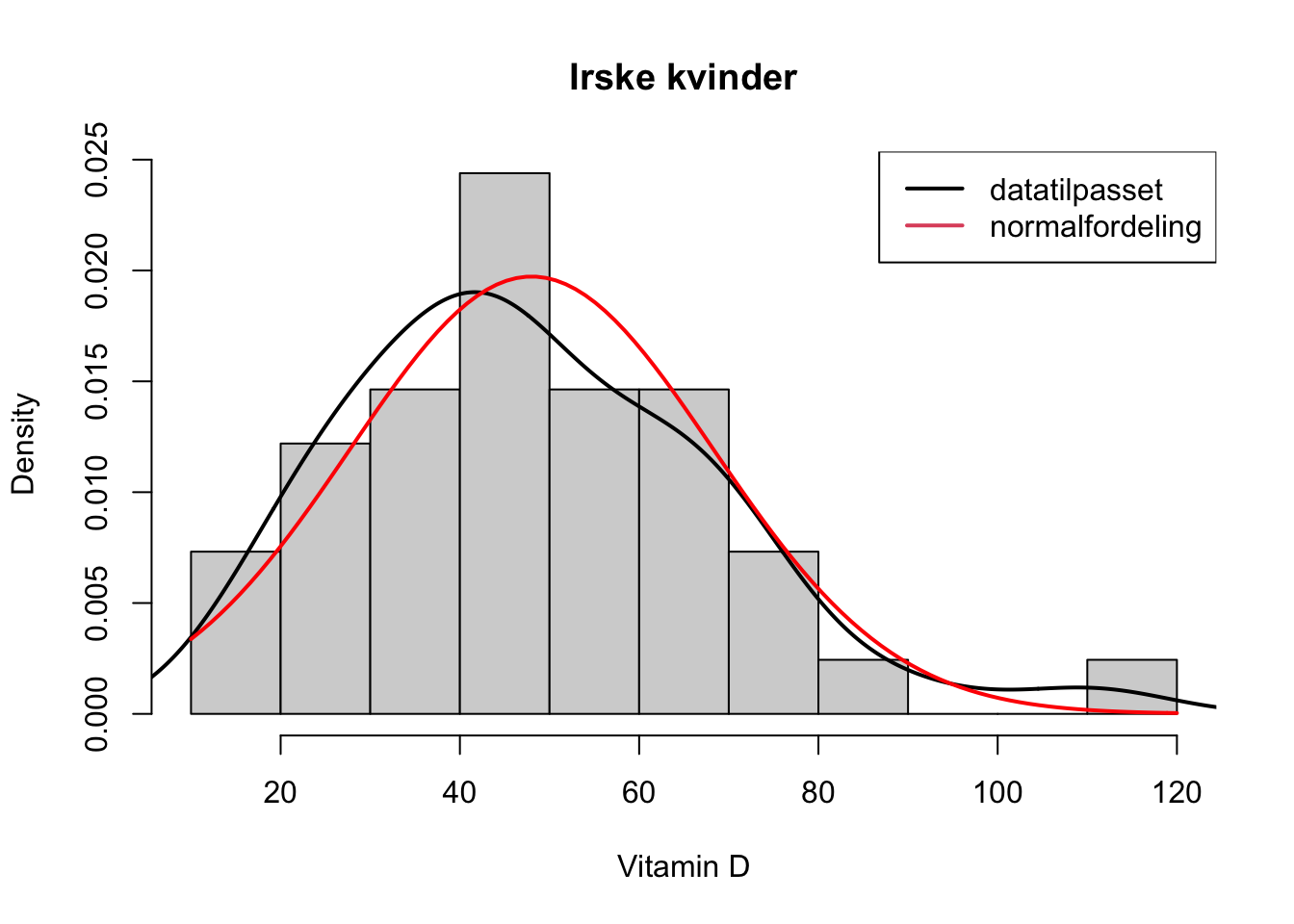
2.2 Boxplot
Eksempel: Vitamin D (uge 1)
boxplot( vit$vitd ~ vit$country, xlab='Land', ylab='Vitamin D')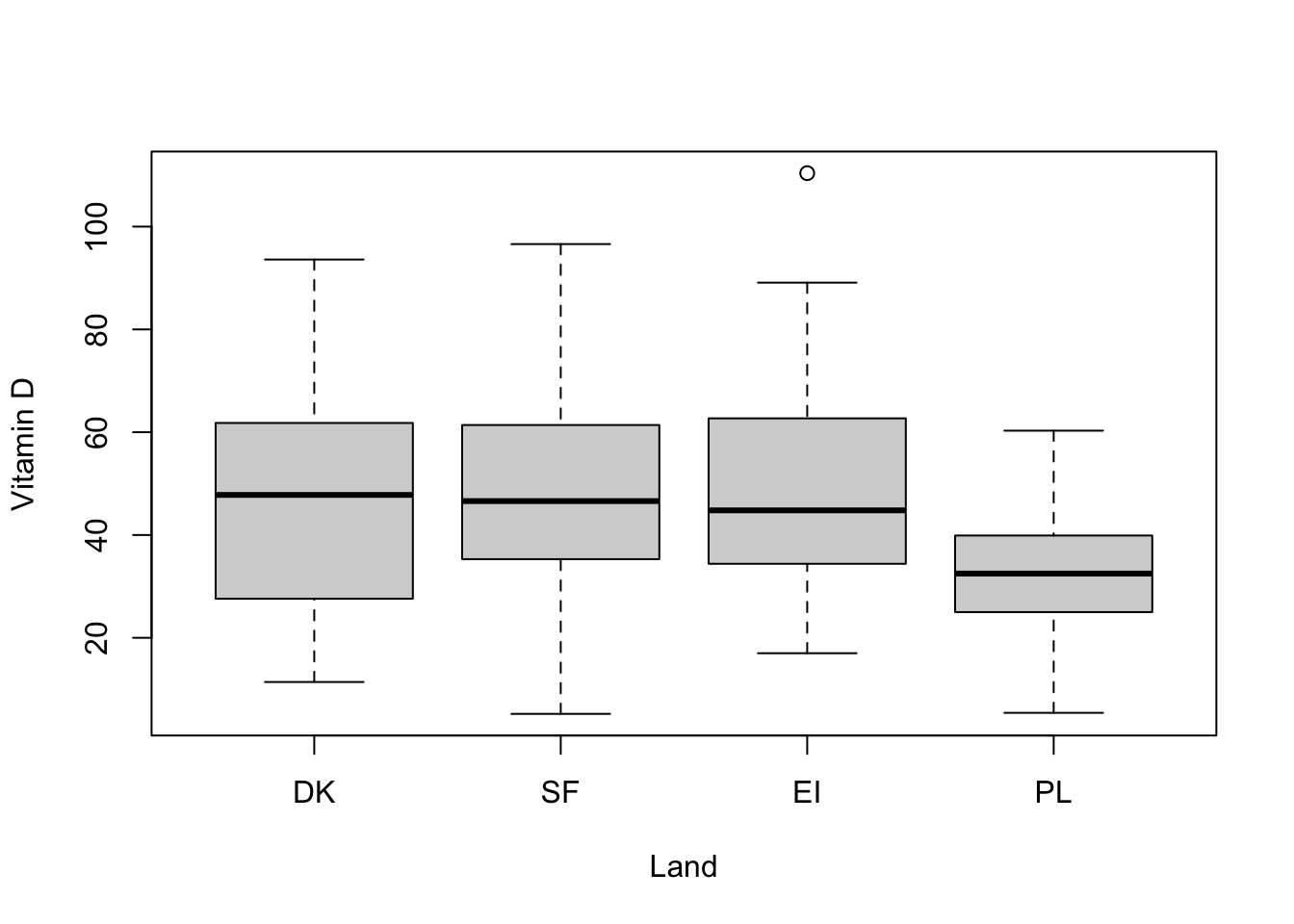
Teknisk: R angiver ikke selv middelværdierne, så dem kan man selv sætte på med points(), som fungerer ligesom lines (beskrevet i video om grafik i R-intro, bortset fra at den tilføjer punkter i stedet for linjer. Bemærk at boxplot nummererer boksene 1-4, så vi skal plotte means (y-værdierne) som funktion af x-værdierne 1-4:
boxplot( vit$vitd ~ vit$country, xlab='Land', ylab='Vitamin D', col='lightgray' )
points(1, mean(vit$vitd[vit$country=='DK']), pch=5)
points(2, mean(vit$vitd[vit$country=='SF']), pch=5)
points(3, mean(vit$vitd[vit$country=='EI']), pch=5)
points(4, mean(vit$vitd[vit$country=='PL']), pch=5)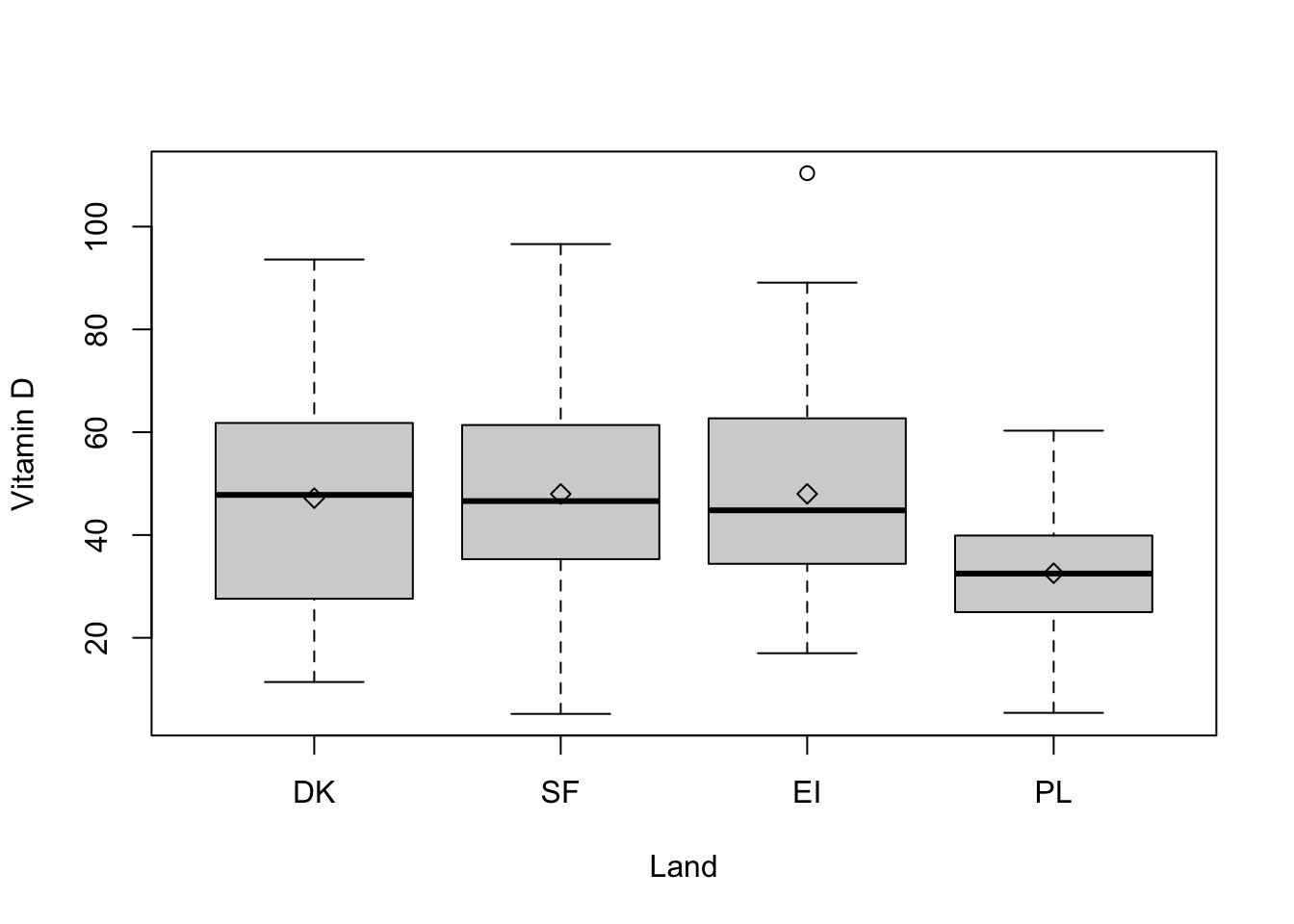
2.3 QQ-plot
Eksempel: Vitamin D (kun irske kvinder, uge 1)
qqnorm( vitEI$vitd )
qqline( vitEI$vitd )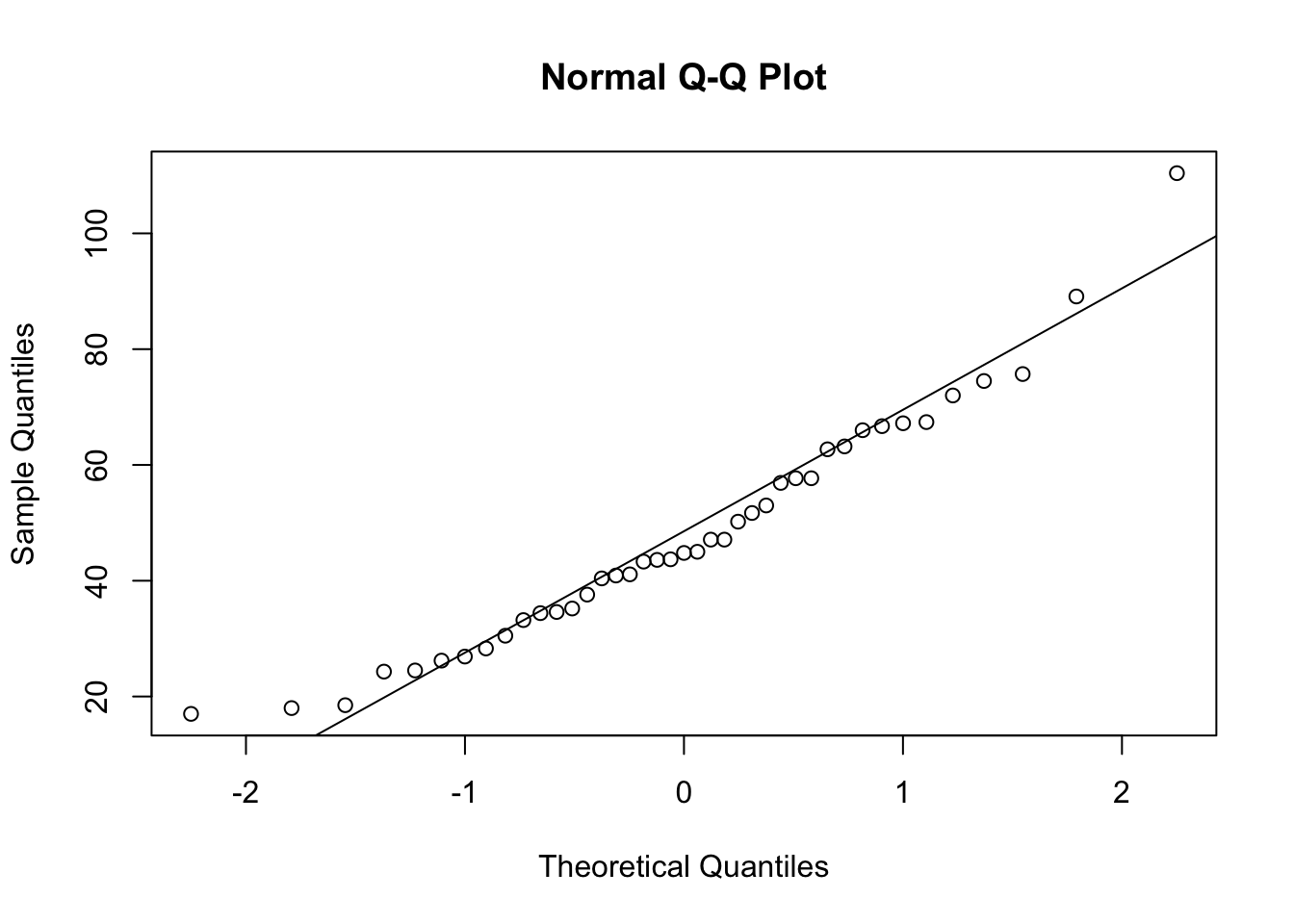
2.4 Scatterplot med linjer
Eksempel: Slagvolumen målt ved to metoder (parrede målinger, uge 1)
Med abline() tilføjer vi en ret linje med intercept i a=0 og hældning b=1 (heraf navnet AB line) til et scatter plot af SV vs MF:
plot( ms$mf ~ ms$sv, pch=19, xlab='SV', ylab='MF',
xlim=c(40,135), ylim=c(40,135))
abline( a=0, b=1, lwd=2, col='red' ) 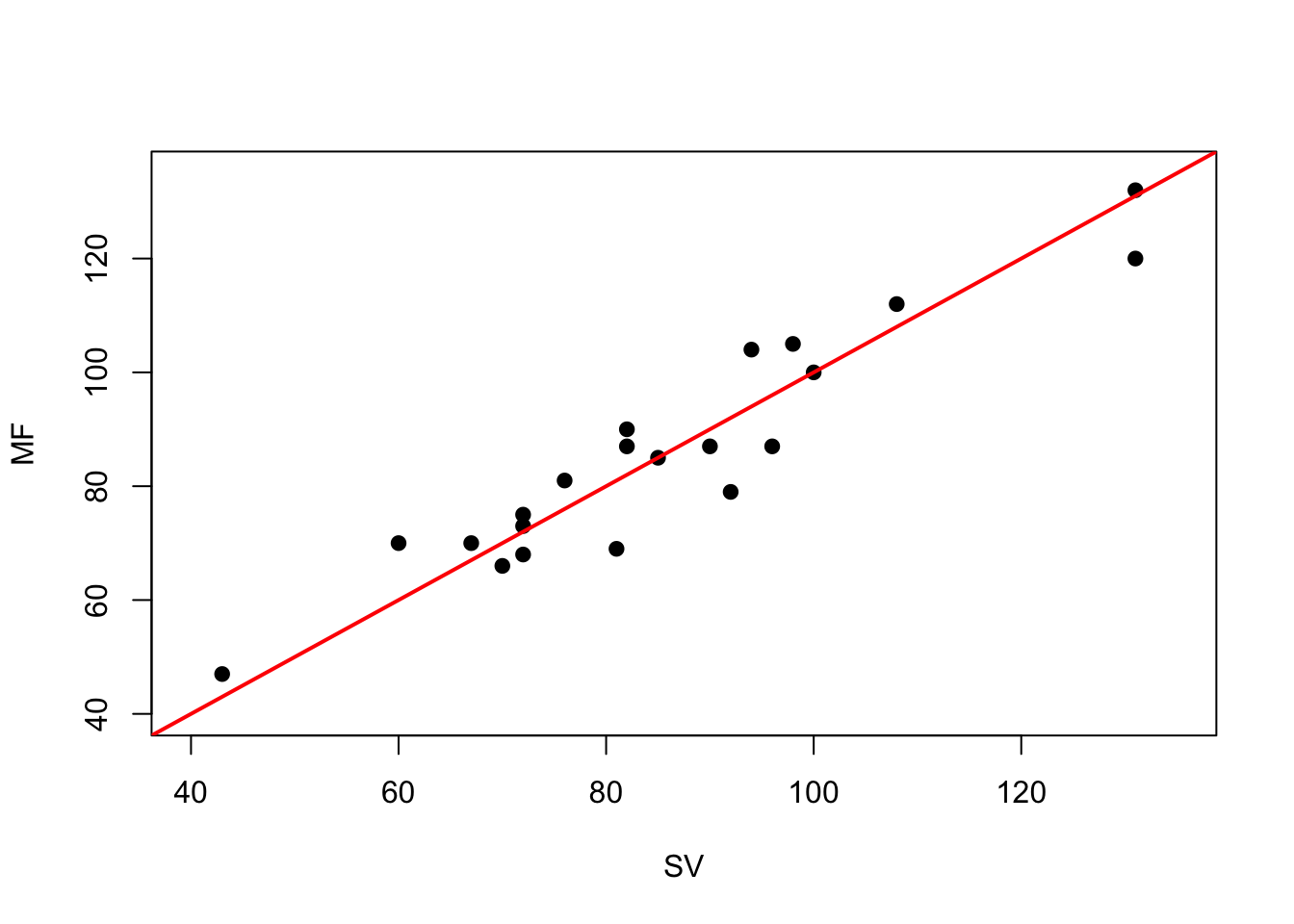
2.5 Spaghettiplot
Eksempel: Slagvolumen målt ved to metoder (parrede målinger, uge 1)
Et spaghettiplot kræver at data er på langt format, dvs at alle målingerne står i én søjle (her med navn vol), mens en anden forklarende variabel metode angiver hvilken metode der er tale om. Data i langt format indlæses fra datafilen mf_sv_lang.csv:
lang <- read.csv('http://publicifsv.sund.ku.dk/~sr/BasicStatistics/datasets/mf_sv_lang.csv')
head(lang) vol metode id
1 47 mf 1
2 66 mf 2
3 68 mf 3
4 69 mf 4
5 70 mf 5
6 70 mf 6tail(lang) vol metode id
37 100 sv 16
38 94 sv 17
39 98 sv 18
40 108 sv 19
41 131 sv 20
42 131 sv 21Spaghettiplot laves med pakken lattice - den skal først installeres med
install.packages('lattice')install-linjen køres kun en gang, så den kan du slette fra dit script, når den er kørt.
Hver gang du på ny starter R og ønsker at bruge pakken, skal du gøre indholdet tilgængeligt med
library(lattice)ellers kan R ikke finde de funktioner, som ligger i pakken.
Vi kan nu benytte xyplot() til at lave spaghettiplot, her skal man dog først definere den variabel man vil have på x-aksen som en faktor, ellers strejker R:
lang$metode <- factor(lang$metode)
xyplot( vol~metode, groups=id, data=lang, type="b", col='darkblue', pch=19)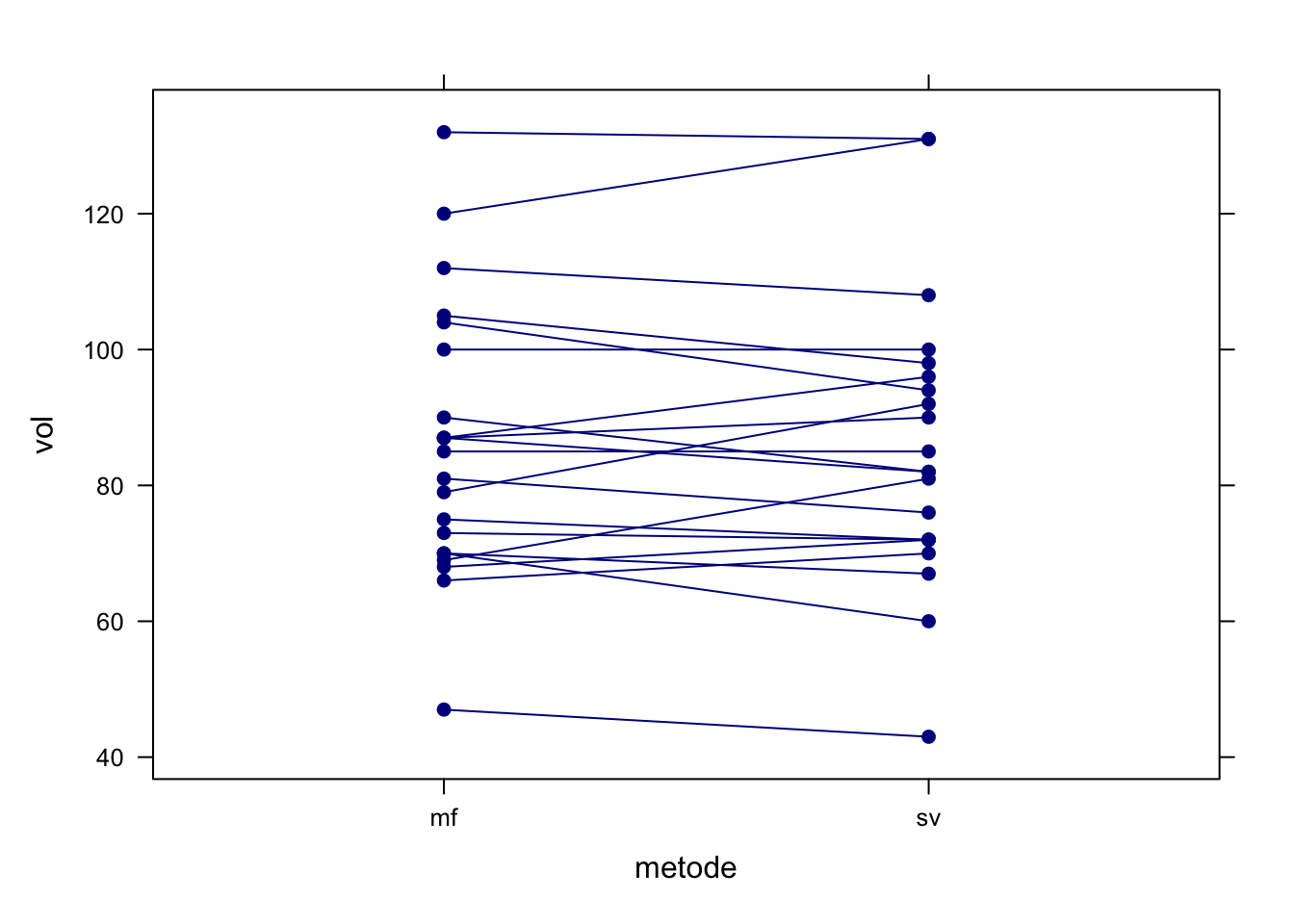
Vi plotter altså volumen som funktion af metode og specificerer at målingerne skal forbindes svarende til id-variablen (groups=id), type='b' (Both) angiver at vi ønsker både linjer og punkter.
2.6 Bland-Altman plot
Eksempel: Slagvolumen målt ved to metoder (parrede målinger, uge 1)
Plot af differens vs gennemsnit
ms$dif <- ms$mf-ms$sv
ms$gns <- 0.5*(ms$mf+ms$sv)
head( ms ) mf sv dif gns
1 47 43 4 45.0
2 66 70 -4 68.0
3 68 72 -4 70.0
4 69 81 -12 75.0
5 70 60 10 65.0
6 70 67 3 68.5plot( ms$dif ~ ms$gns, pch=19, ylim=c(-15,15), xlab='Gennemsnit',
ylab='Differens', main='')
abline( h=0, col='gray' ) 
Tilføj selv LOA, se R-script uge 1.
2.7 3D-plot
Eksempel: Fødselsvægt (Secher), uge 6
Med tre kvantitative variable (to kovariater og en responsvariabel) kan vi illustrere data i et 3D-scatterplot.
Med funktionen scatterplot3d kan man lave 3D-plots, hvor man angiver hvilke variable, der skal være ud af hver akse, som vi kender det fra den almindelige plot-funktion - nu skal der bare angives tre akser.
Først installeres og indlæses pakken scatterplot3d:
install.packages("scatterplot3d")library(scatterplot3d)I eksemplet er vaegt responsvariablen hvorfor vi vælger at sætte den som tredje argument (lodret akse):
scatterplot3d( secher$ad, secher$bpd, secher$vaegt, pch = 20, highlight.3d = TRUE)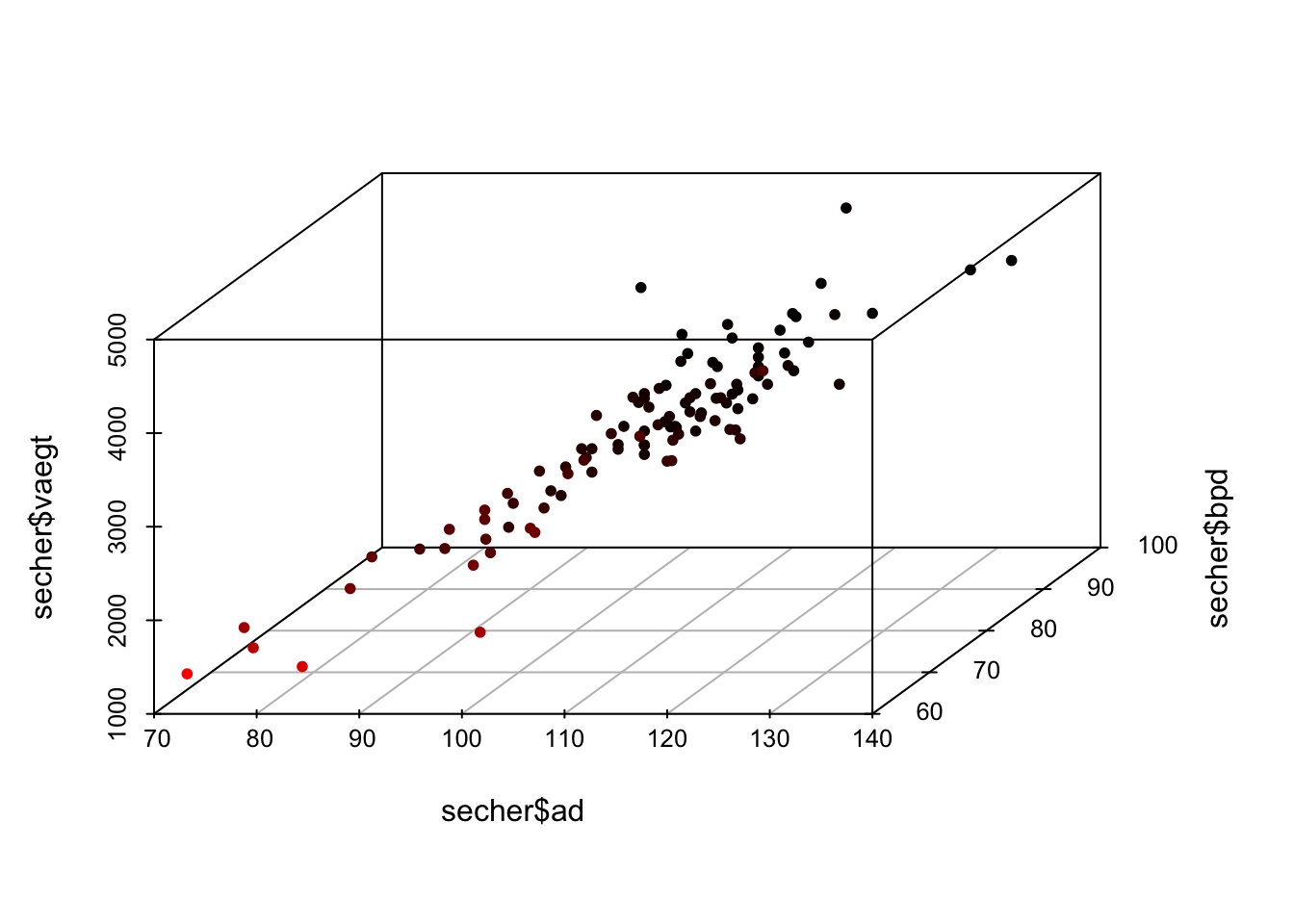
Der findes et hav af ekstra argumenter, kør eventuelt ?scatterplot3d for at se hvilke parametre man kan ændre i plottet. Her har vi roteret figuren 45 grader med uret (angle=-45) og tegnet lodrette linjer for at forbinde punkterne (type="h"):
scatterplot3d( secher$ad, secher$bpd, secher$vaegt, pch = 20, highlight.3d = TRUE,
angle=-45, type = "h")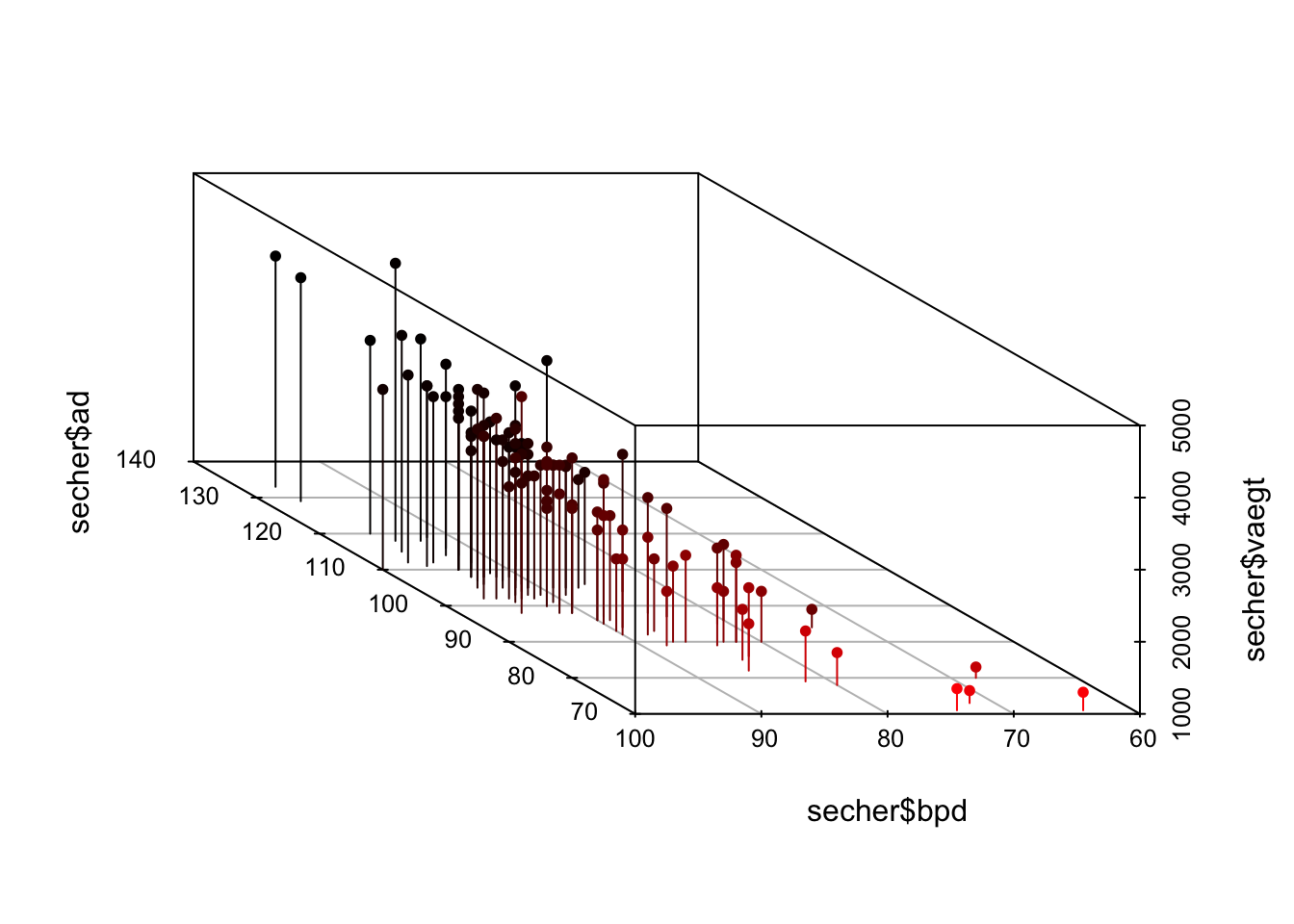
2.8 Bubble plot
Eksempel: Fødselsvægt (Secher), uge 6
En anden måde vi kan visualisere sammenhængen mellem fødselsvægt og de forklarende variable er ved et 2D-plot med ggplot, hvor størrelsen og farven af punkterne indikerer fødselsvægten. At lave et ggplot er lidt anderledes end at lave et plot med plot-funktionen eller scatterplot3d. Først angives den overordnede struktur af figuren i ggplot(), hvorefter man kan tilføje elementer eller instillinger med +.
I eksemplet nedenfor er plottet opbygget på følgende måde:
I
ggplotangives først hvilket datasæt, der benyttes i figuren, hersecher. Herefter angives iaes()hvad der skal være ud af x-aksen og y-aksen, og at størrelsen skal afspejlevaegt.Med
geom_pointtilføjer vi observationerne i datasættet som punkter. Iaes()angiver vi desuden, at farven af punkterne skal afspejlevaegt.I
scale_size_continuousjusterer vi variationen i størrelser af markører, der er mulige, medrangeargumentet.
install.packages("ggplot2")library(ggplot2)
ggplot(secher, aes( x = bpd, y = ad, size = vaegt)) +
geom_point( aes(colour = vaegt)) +
scale_size_continuous( range = c(0.5, 12)) +
theme(text=element_text(family="Times New Roman", size=12),
axis.text = element_text(size = 12,colour="black"))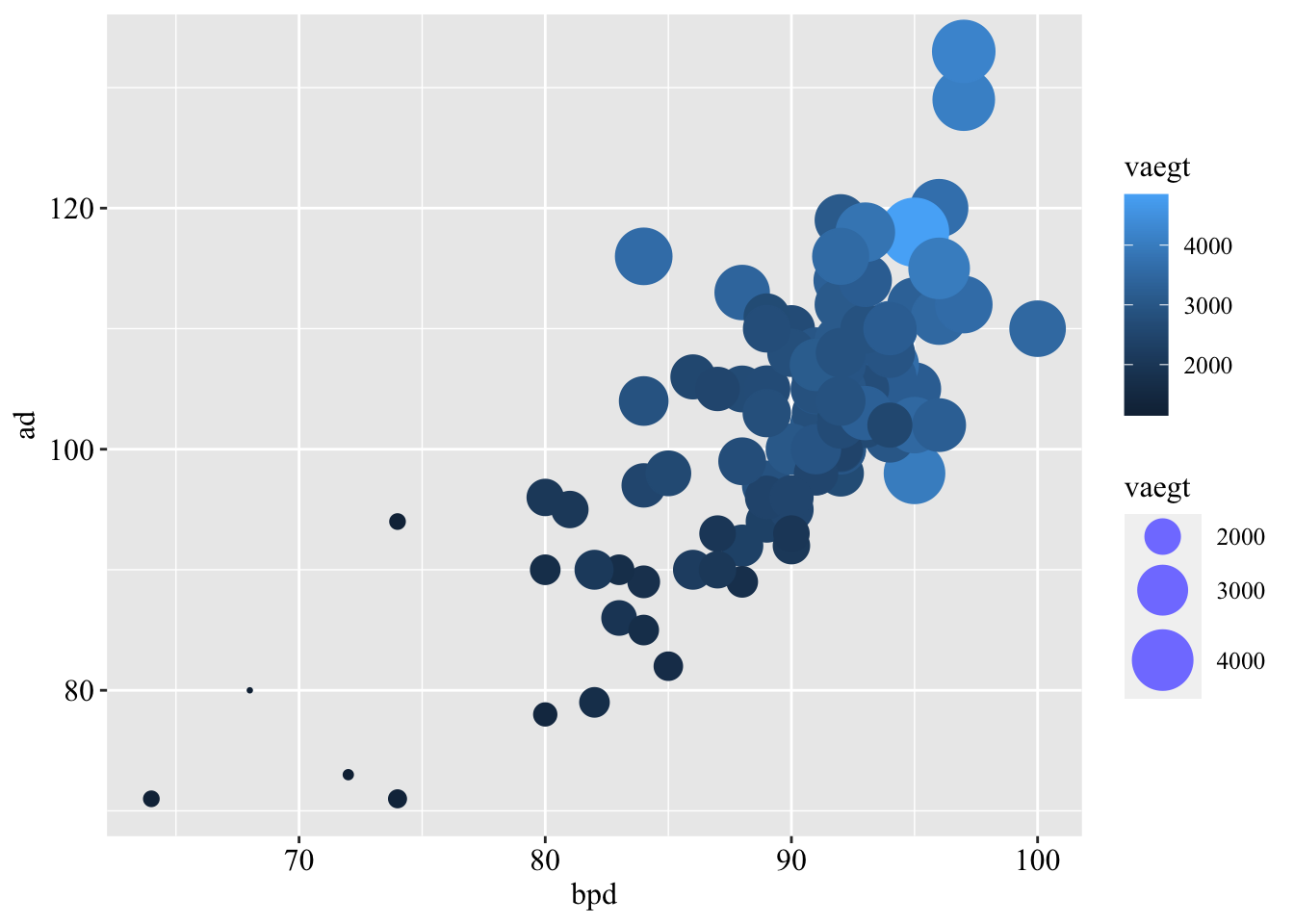
NB: Jeg har efter opdatering af min computer problemer med mine fonte og farver, så plottet ser ikke ud som det plejer for mig og jeg er nødt til at specificere ovenfor, at jeg ønsker at labels på akserne skal skrives i sort etc … Det er ikke sikkert at det virker lige så skørt hos dig, måske du kan klare dig med færre linjer a la nedenstående.
Hvis ggplot er nyt for dig, kan det være en fordel først at køre ggplot-delen og herefter køre ggplot- og geom_point-delene osv. for at får en fornemmelse af hvordan, der bliver bygget videre på figuren, når man tilføjer flere detaljer med +:
ggplot(secher, aes( x = bpd, y = ad, size = vaegt))
ggplot(secher, aes( x = bpd, y = ad, size = vaegt)) +
geom_point( aes(colour = vaegt))
ggplot(secher, aes( x = bpd, y = ad, size = vaegt)) +
geom_point( aes(colour = vaegt)) +
scale_size_continuous( range = c(0.5, 12))
# etcDet kan virke omstændigt at bruge ggplot i begyndelsen, men det kan være et rigtig godt og fleksibelt redskab til at lave mange forskellige figurer, som man gerne vil have dem.
Se denne side for tilpasning af plottet / flere eksempler.